Nature:开创性空间多组学技术!首次实现同一切片DNA甲基化和转录组的空间联合分析
时间:2025-09-09 12:13:53 热度:37.1℃ 作者:网络
DNA甲基化可以不改变基因序列调控基因表达,并随细胞分裂遗传,在癌症、衰老等复杂疾病的发生发展中起着关键作用。目前,DNA甲基化是研究最透彻的表观遗传修饰之一,虽然单细胞甲基化分析已取得较大进展,但仍缺乏完整组织的空间信息,这限制了对组织发育和疾病进展过程中基因调控的理解。
空间多组学技术是一种综合性的研究方法,能够结合原位组织的多组学信息和空间信息,全面了解原位组织的生物复杂性。但已有的空间多组学方法仅限于组蛋白修饰、染色质可及性、转录组和选定的蛋白质panel,DNA甲基化的直接空间映射还没有实现。
近日,美国宾夕法尼亚大学研究团队在Nature发表文章“Spatial joint profiling of DNA methylome and transcriptome in tissues”,首次在组织原位实现了DNA甲基化与转录组的联合分析。研究团队报道了一种开创性的空间多组学技术spatial-DMT,可以在同一组织切片上对全基因组DNA甲基化和转录组进行空间联合分析。该技术填补了DNA甲基化分析的空白,拓展了空间组学的应用范围,有助于更全面地了解组织生物学在发育和疾病中的作用,也为发现甲基化相关的分子标志物、深入理解疾病机制以及推动转化应用奠定了基础。

主要研究内容
spatial-DMT技术
spatial-DMT结合了微流控原位条形码技术、胞嘧啶脱氨基转换和高通量NGS技术,通过在组织切片中同时捕获DNA和RNA分子,并加上空间条形码,从而直接在原组织中实现空间甲基组化和转录组分析。
具体而言,该工作流程首先利用盐酸处理固定的冷冻组织切片,提高DNA甲基化分析的可及性;然后进行Tn5转座和逆转录,插入通用接头;组织基因组片段和cDNA依次连接空间条形码,两组空间条形码在微流控通道中相互垂直流动,在目标区域形成2500个空间条形码组织像素的二维网格,分辨率低至10μm,近似单细胞分辨率;分离cDNA和gDNA,cDNA通过模板切换后制备cDNA文库;gDNA通过酶促甲基测序(EM-seq)转换和连接处理,之后构建文库。为最大限度地减少DNA损伤,研究团队创新性利用温和的EM-seq进行转化,在减少DNA损伤的同时,实现了未修饰胞嘧啶向尿嘧啶的高效转化(转化效率>99%)。
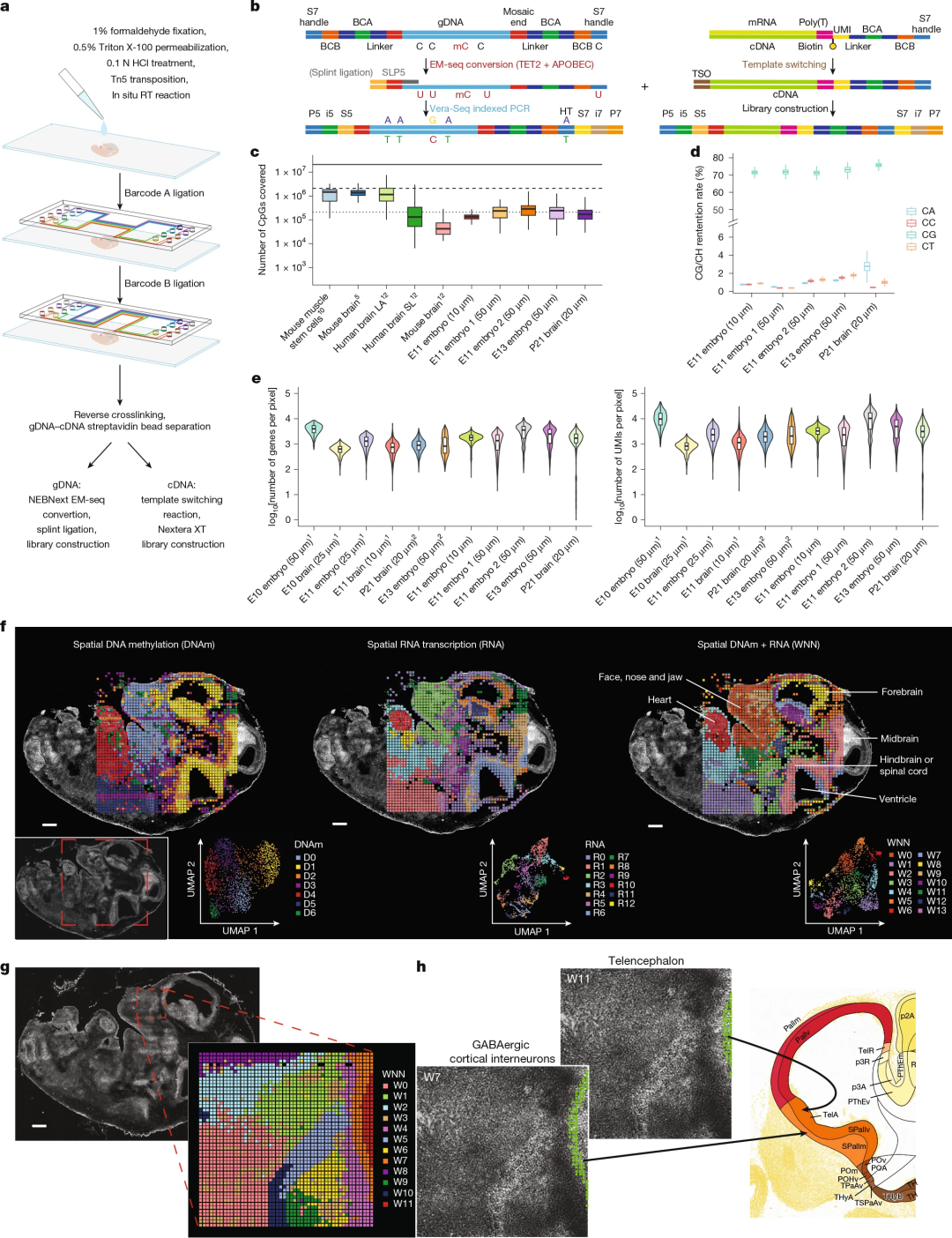
图1.spatial-DMT工作流程和组织中DNA甲基化和转录组共谱分析概述。
spatial-DMT技术验证
为了验证spatial-DMT技术在复杂组织中共同分析DNA甲基化和转录组的性能,研究团队分析了胚胎11天(E11)和13天(E13)的小鼠胚胎以及出生后21天(P21)的小鼠大脑。其中小鼠胚胎以两种像素分辨率绘制:50μm用于头部和上半身,10μm用于放大面部和前脑区域。对出生后小鼠大脑以20μm像素分辨率进行分析。
结果显示,在匹配身体部位的E11胚胎重复图谱中,DNA甲基化与RNA表达显示出高度一致性,表明spatial-DMT具有可重复性。DNA甲基化分析对E11、E13和P21脑样本产生了28 - 39亿个原始reads,平均每像素覆盖136,639-281,447个CpGs,与此前对小鼠胚胎和大脑样本的单细胞DNA甲基化研究相当。对无甲基化连接子序列的分析表明,超过99%的胞嘧啶被成功转化,进一步证实了spatial-DMT的转化效率。CpG覆盖的基因组分布评估显示,spatial-DMT产生了准确和公正的基因组DNA甲基化分析。同时,spatial-DMT从同一组织切片中生成了高质量的RNA数据,可以直接比较转录活性和表观遗传状态。此外,在DNA甲基化文库中没有检测到RNA污染的证据。
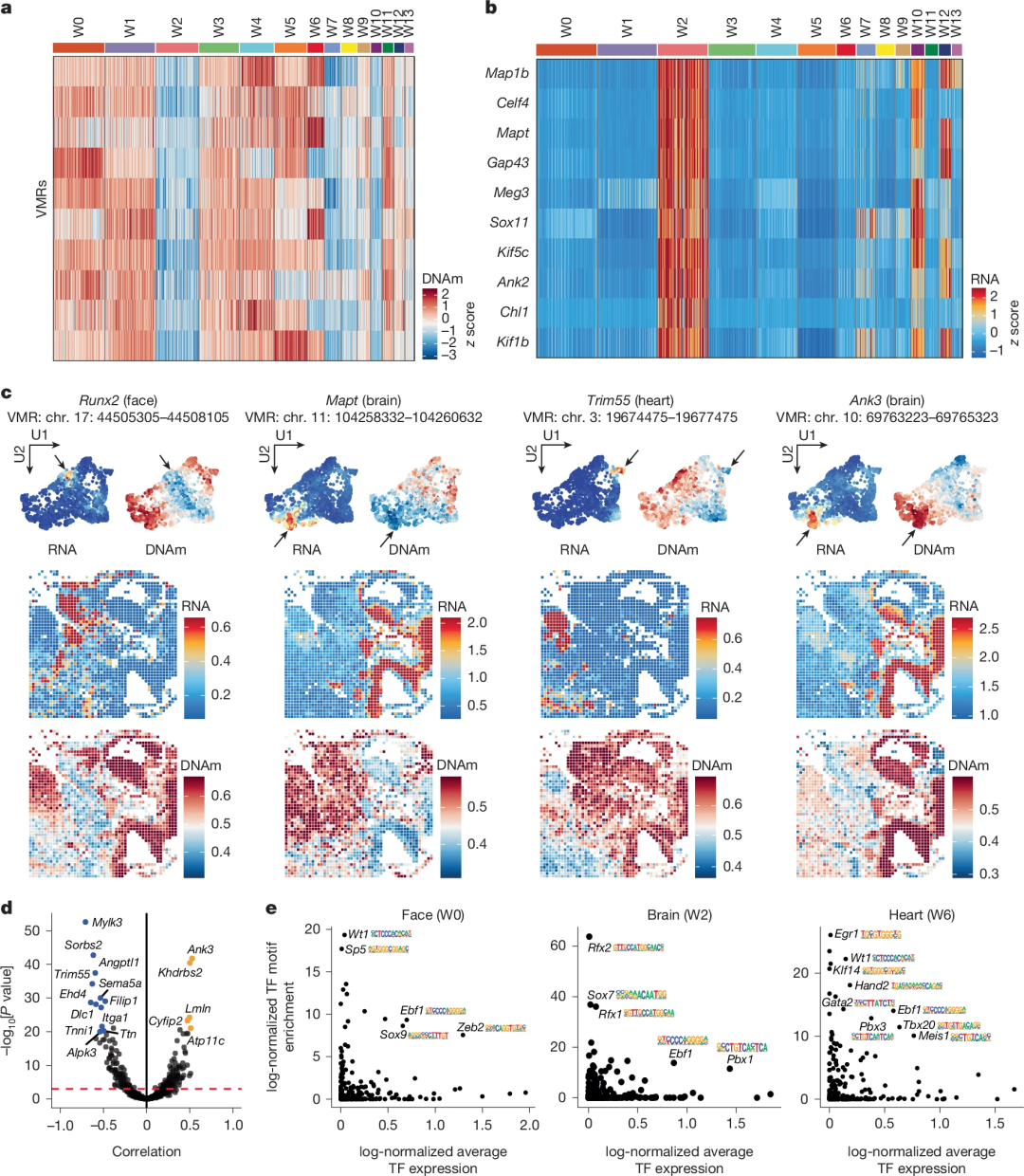
图2.小鼠E11胚胎DNA甲基化及基因表达分析。
研究团队利用spatial-DMT首次绘制了E11小鼠胚胎DNA甲基化和基因表达的空间组织图谱。在该分析中,DNA甲基化和转录组可以独立定义细胞身份,每种模式都捕获了细胞身份的不同但互补的方面,并通过WNN分析整合在一起,产生了分辨率更高的集群,突出了空间多组学整合在解决单模态分析遗漏的细胞异质性方面的优势。
研究分析了小鼠胚胎大脑(W2)、颅面(W0)和心脏(W6)特异性可变甲基化区域和邻近基因表达(图2),发现空间簇特异性基因表达通常与邻近可变甲基化区域的低DNA甲基化相关,例如在颅面区域(颌骨和上鼻)、脑、脊髓以及心脏(图2c)。同时,研究也观察到部分基因的RNA表达与相关可变甲基化区域的甲基化水平呈正相关。该结果揭示了DNA甲基化在转录调控中的复杂作用。此外,研究团队使用10μm像素的微流控芯片绘制了接近单细胞分辨率的小鼠大脑空间图谱,以精确解析小鼠E11胚胎颅面和前脑区转录调控的精细结构,揭示了胚胎神经和感觉系统的形成过程。以上结果强调了spatial-DMT在解决复杂解剖结构和捕获时空动态方面的能力。
通过对小鼠E11和E13胚胎的spatial-DMT分析,研究团队在空间和时间尺度上研究了DNA甲基化、基因表达及其相互作用的动态,揭示了序列、细胞类型和区域特异性甲基化介导的转录调控的细节。例如,spatial-DMT生成的组织空间图谱数据可以更精确地将发育过程中的上调基因和通路激活时间定位到胚胎发育的特定阶段、组织位置,并揭示它们的表观遗传调控机制。
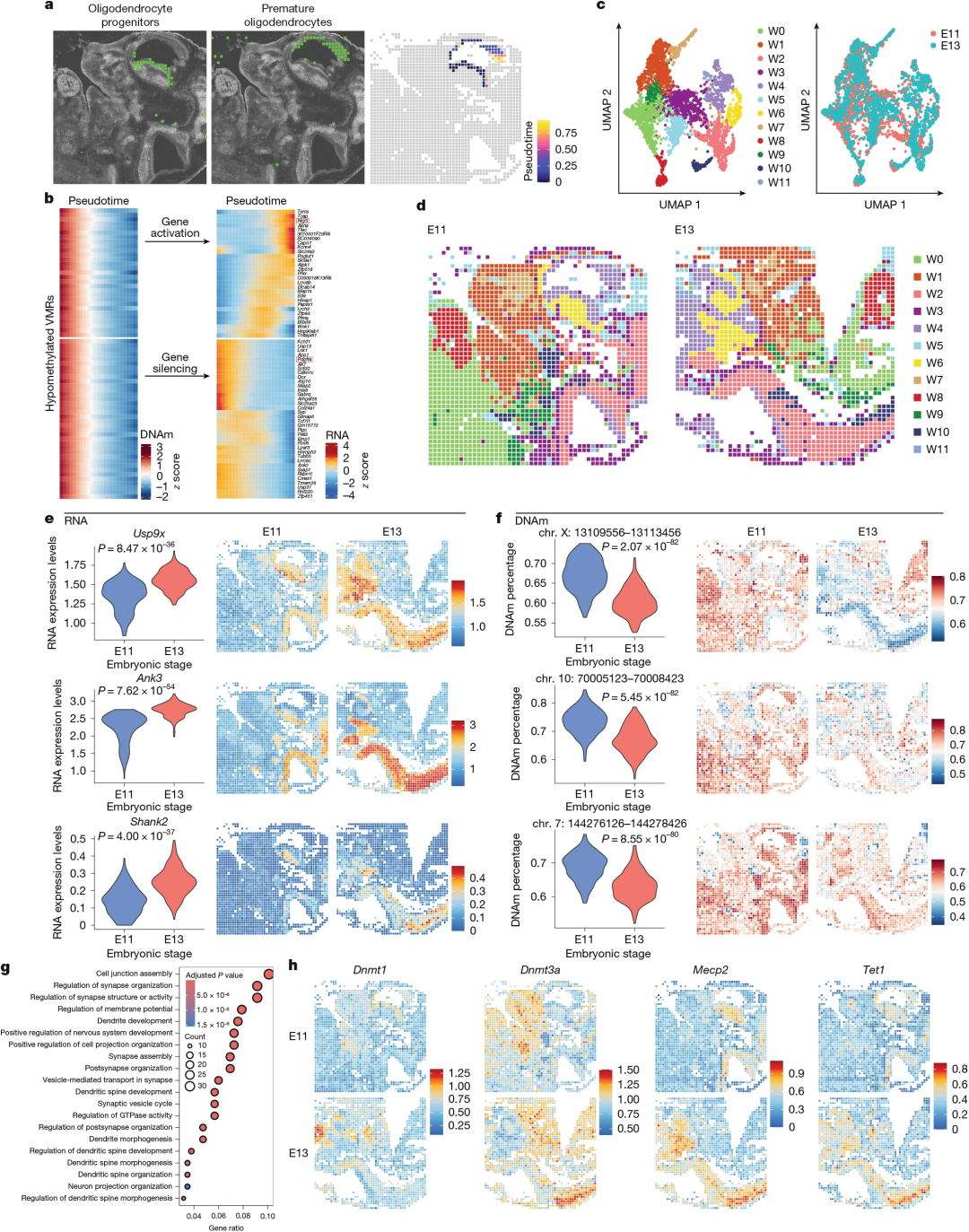
图3.胚胎发育过程中DNA甲基化和RNA转录的时空动态。
此外,spatial-DMT还能识别转录组无法区分的表观亚群,分析不同组织微环境中微妙的表观遗传异质性,例如与颅面和心脏形态发生相关的调控元件。(图4)
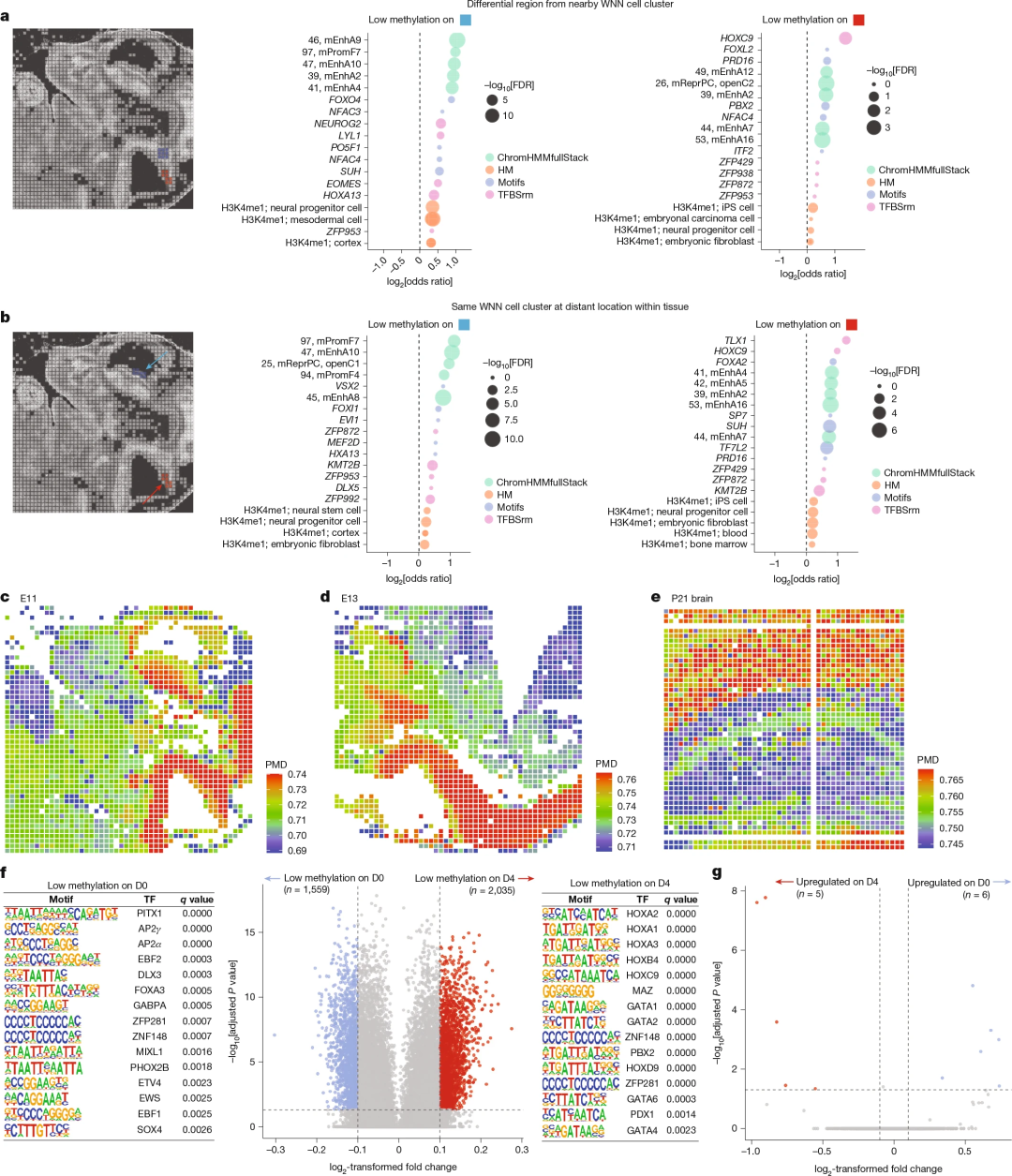
图4.spatial-DMT分析不同组织微妙的区域特异性表观遗传变异。
结 语
研究团队展示的开创性空间多组学技术spatial-DMT,可以在同一组织切片中进行DNA甲基化和RNA表达的同步分析,并无需组织解离,可完整保留细胞的空间位置及组织微环境。研究团队创新性的利用微流控芯片进行了原位组织的二维网格划分,并标记空间条形码,还优化了DNA甲基化的捕获,使用温和的EM-seq进行转化,适用于FFPE样本。经验证,该技术可产生高质量数据,且具可重复性和一致性。
研究团队表示,未来spatial-DMT可能会结合更多的分子检测,包括染色质构象(HiC)、可及性(ATAC-seq)、组蛋白修饰(CUT&Tag)、代谢组(质谱成像)和表面蛋白(CITE-seq),以提高基因原位调控机制的分辨率。
总之,spatial-DMT为高分辨率、无偏倚的全基因组甲基化分析提供了一种具有成本效益的策略,推进了空间组学分析和甲基化生物标志物的发现,为表观遗传研究提供了全新框架,有助于对疾病机制的研究和临床转化。
原文信息:
Lee, C.N., Fu, H., Cardilla, A. et al. Spatial joint profiling of DNA methylome and transcriptome in tissues. Nature (2025). https://doi.org/10.1038/s41586-025-09478-x


