Genome Biol:曹亚南/孙洁林团队开发知识图谱增强框架KEGNI推断基因调控网络
时间:2025-09-26 12:11:47 热度:37.1℃ 作者:网络
在每个细胞中,基因通过基因调控网络(Gene regulatory network, GRN)相互协作,在维持细胞稳态、调控发育进程以及响应外界刺激等生物过程中发挥关键作用。阐明细胞内基因与调控因子之间的互作关系,是理解细胞正常功能及病理状态分子机制的重要基础。解读这些基因调控网络是我们理解生命奥秘和疾病机制的关键。近年来,单细胞RNA测序(scRNA-seq)技术的发展为细胞类型特异的GRN分析提供了数据基础,但是如何利用海量数据实现准确推断,仍是一个巨大挑战。现有的方法多依赖于基因共表达关系或多组学数据整合,仍面临假阳性率高、多组学数据获取和整合受限和先验知识利用不足等问题。
近日,上海交通大学医学院附属瑞金医院曹亚南研究员和上海交通大学孙洁林教授团队合作开发了一个名为KEGNI的人工智能新方法进行GRN推断,在Genome Biology杂志发表了“KEGNI: knowledge graph enhanced framework for gene regulatory network inference”论文。团队发布了一个知识图谱增强的基因调控网络推断框架KEGNI(Knowledge graph-Enhanced Gene regulatory Network Inference),通过整合已有生物学知识从单细胞RNA测序数据中推断细胞类型特异的基因调控网络,避免了依赖基因共表达关系或非配对多组学数据的方法所存在的局限性。

KEGNI结合了两种强大策略用于基因调控网络推断。KEGNI的核心是一个“掩码图自动编码器”(Masked graph Autoencoder,MAE),它通过随机遮盖部分基因的表达数据并尝试复原,从而自主、深入地学习基因之间隐藏的调控模式,实现“自我学习”的能力。更重要的是,KEGNI嵌入了知识图谱(Knowledge Graph Embedding,KGE),一个整合了KEGG等数据库中已知基因相互作用关系的数据库。知识图谱模块可为掩码图自动编码器提供了正确信息,确保其学习过程不偏离已有的生物学事实。KEGNI两个模块联合优化基因表达特征重构和知识图谱嵌入,执行“自学”与“名师指导”相结合的多任务学习策略,能够推断出细胞类型特异的基因调控网络(图1)。
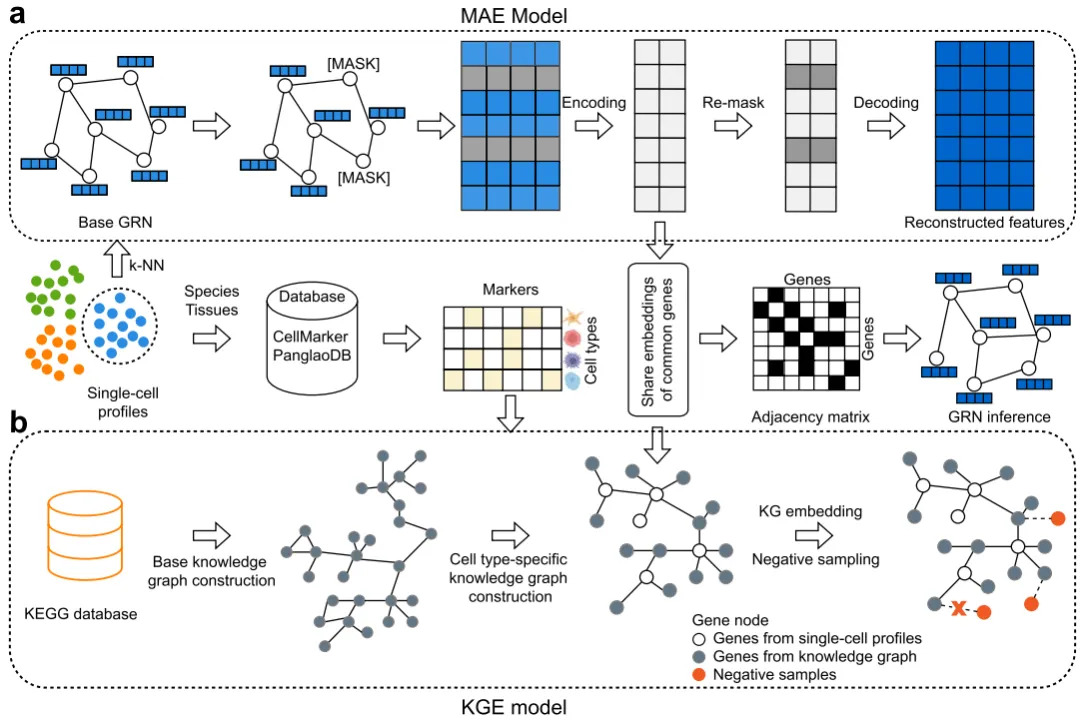
图1. KEGNI框架图
经过多项基准测试结果,和现有基于scRNA-seq进行GRN推断的多种方法比较,KEGNI表现出显著的优势(图2)。与其他方法相比,KEGNI推断出的基因关系更接近已知的生物学事实,精准性更高。KEGNI仅需scRNA-seq数据即可达到优异效果,其性能可与基于成本更高获取更复杂的配对scRNA-seq和scATAC-seq数据的方法相媲美,降低了研究的数据门槛,具有更广的适用性。
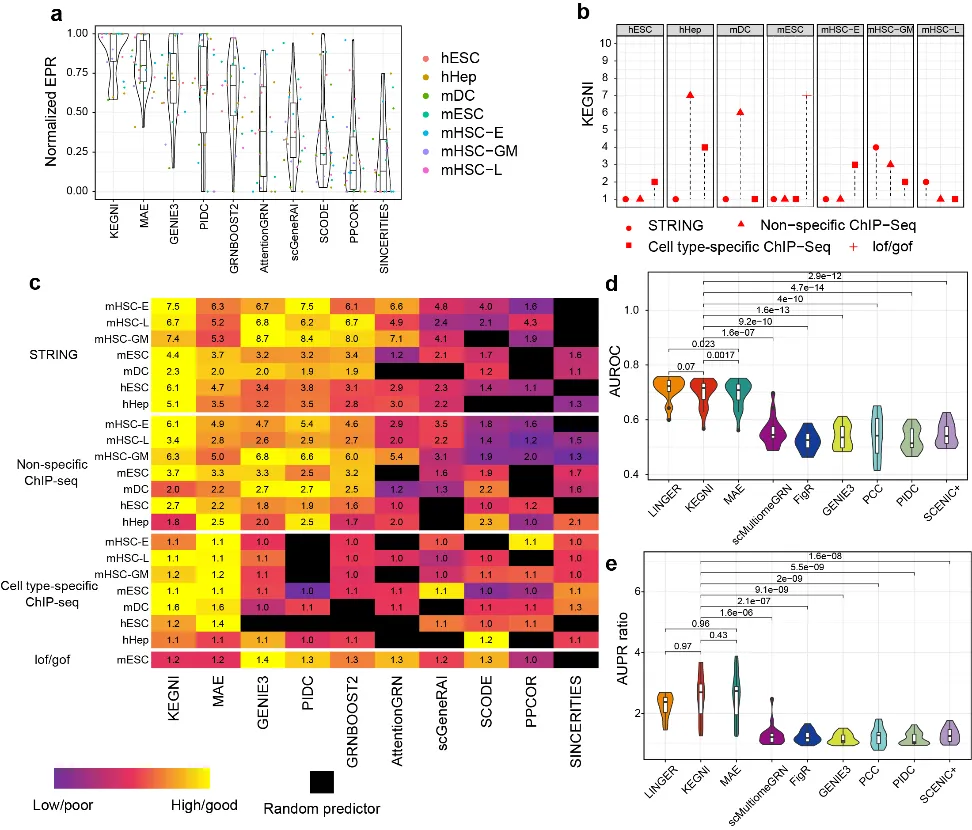
图2. KEGNI与基准方法的比较
另外,研究结果显示KEGNI结果的生物学意义可解释性强,证明引入外部知识能够有效提升调控建模的生物学合理性。研究团队用KEGNI成功识别了在高脂饮食小鼠胰岛β细胞中起关键作用的驱动基因,揭示了不同细胞状态下的调控差异,还验证了KEGNI在小胶质细胞中推断转录因子及非转录因子的调控基因的有效性和准确性,证实了KEGNI在生物学意义解析中的可靠性(图3)。
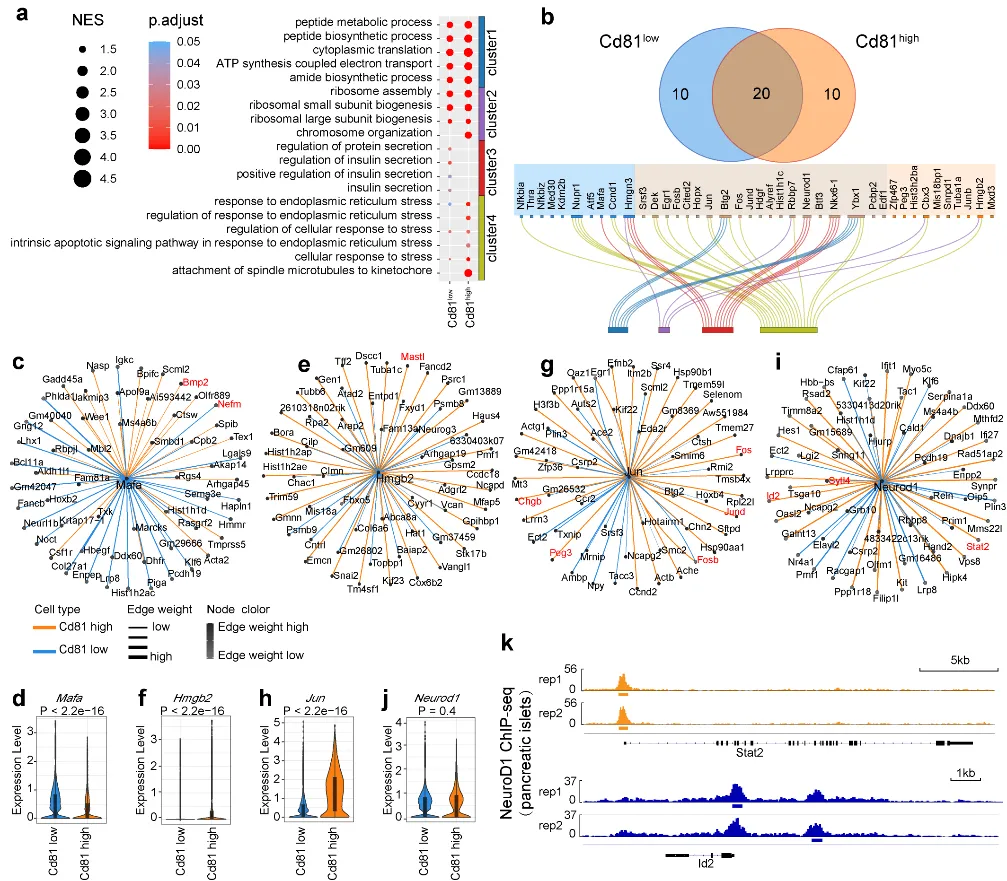
图3. KEGNI在识别驱动基因与调控差异分析中的应用
综上所述,KEGNI通过整合先验知识与scRNA-seq数据实现了细胞类型特异性的基因调控网络推断,显著提升了准确性和模型可解释性。KEGNI是一个具有良好泛化能力的框架,其中掩码图自动编码器和知识图谱嵌入模块可以独立配置。掩码图自动编码器模块仅依赖scRNA-seq数据,无需先验知识,具备更广泛的适用性;而知识图谱嵌入模块所使用的知识图谱也不局限于现有先验数据库,可根据研究需求引入包括miRNA、lncRNA等非编码分子在内的多种数据信息,以适应不同的任务。
上海交通大学李鹏笑博士,李林副研究员为论文的共同第一作者,通讯作者为上海交通大学孙洁林教授和上海交通大学医学院附属瑞金医院曹亚南研究员。
原文链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03780-7


