Nature Methods:江瑞教授团队提出国际上首个单细胞表观基因组基础大模型EpiAgent
时间:2025-09-28 12:16:53 热度:37.1℃ 作者:网络
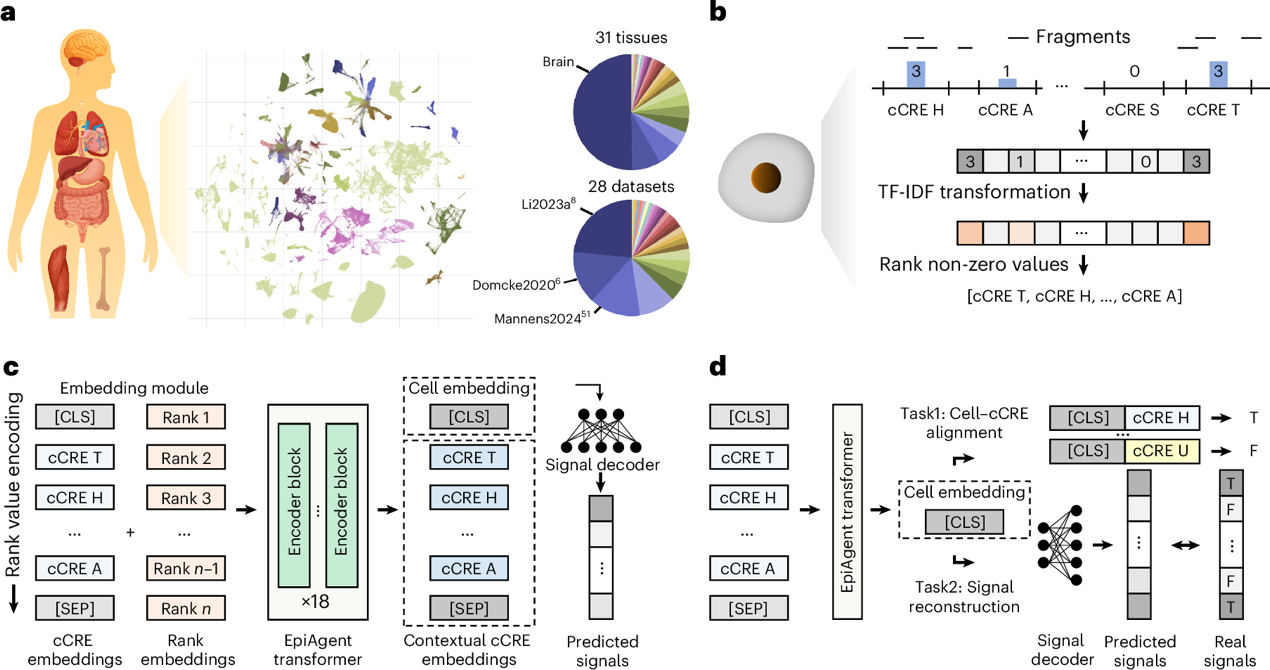
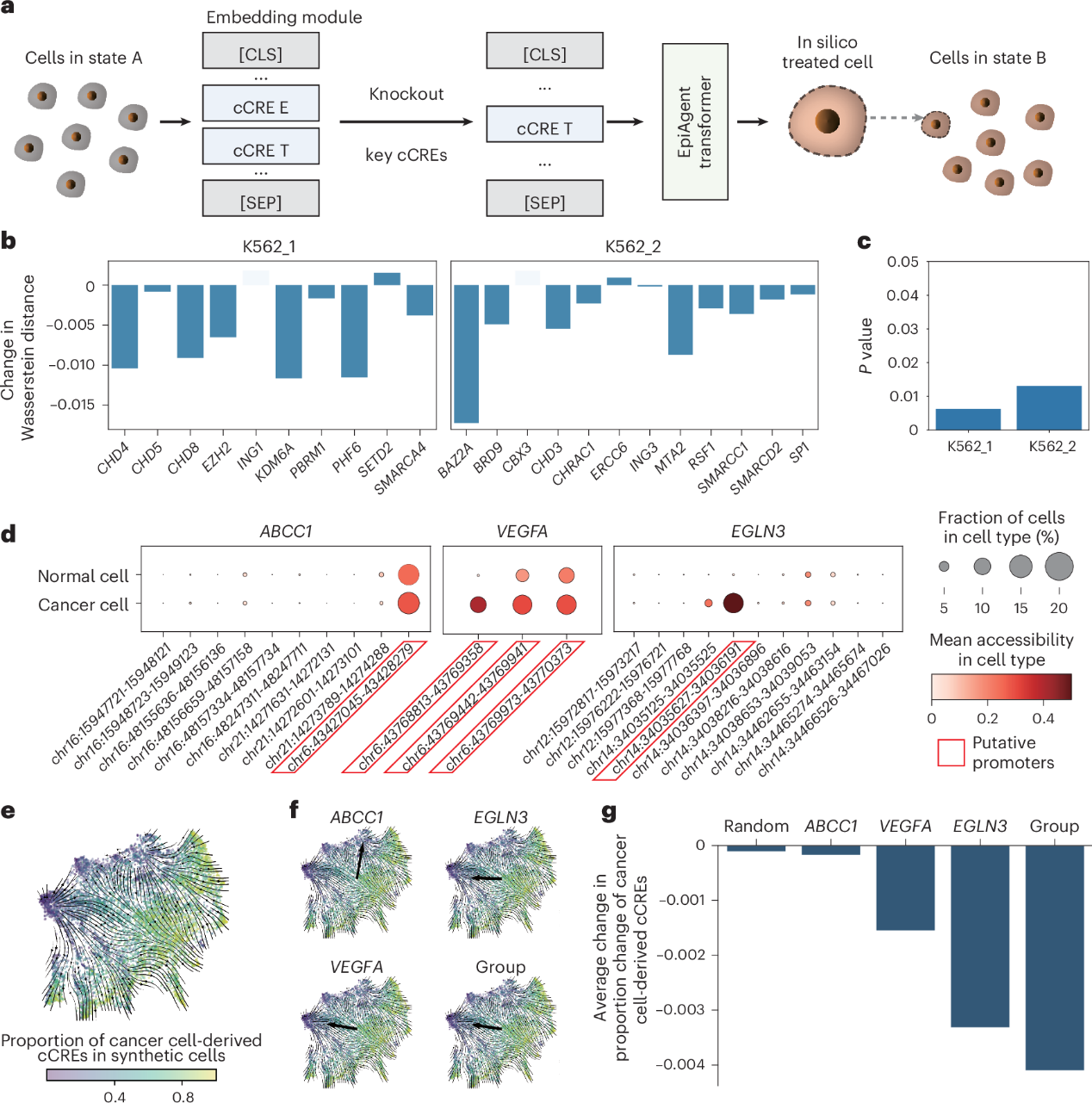
表观基因组通过附加在DNA及相关蛋白质上的修饰精密调节遗传信息的解读和使用,控制基因的转录,是连接DNA序列与人体表型,理解疾病驱动机制的关键桥梁。2015年出现的单细胞染色质开放性测序技术(scATAC-seq)为描绘细胞内的表观基因组景观提供了有力手段,使得在单细胞水平观测基因调控元件的状态成为可能。然而,单细胞染色质开放性数据极为复杂,不仅数据维度高达百万,而且极度稀疏和二值化。这些特点近十年来对下游生物信息学分析提出了严峻挑战,极大制约了这一实验技术的广泛应用。应对这些挑战,清华大学江瑞教授团队提出了国际上首个单细胞表观基因组基础大模型EpiAgent,通过14亿参数的细胞压缩语言模型解决诸多数据分析和建模难题,在细胞图谱构建与映射、扰动响应预测、调控元件虚拟敲除等多个关键领域获得成功应用,开创了表观基因组研究的新范式。 EpiAgent的核心思想是将细胞内由百万染色质开放区域刻画的表观基因组,压缩为一条最多由8192个词元组成的“细胞语句”,通过双向注意力机制在统一语境下从海量数据学习驱动细胞内遗传信息解读的调控规律,以统一的基础模型支撑纷繁复杂的下游应用。如图1a所示,为解决模型构建的数据资源问题,团队耗时两年手工构建了当前最大规模的单细胞染色质开放性数据库Human-scATAC-Corpus(https://health.tsinghua.edu.cn/human-scatac-corpus/),覆盖 31 个组织、28 个公共数据集,包括约 500 万细胞和 350 亿词元(即细胞内开放的调控元件)。为构建细胞语句,EpiAgent仅保留在细胞内开放的调控元件并按其重要性排序,从而实现从海量开放性区域到最多8192个词元的压缩(图1b)。EpiAgent采样三个模块来处理细胞语句(图1c):① 嵌入模块,将开放的调控元件及其排序信息映射到向量空间;② EpiAgent Transformer,采用 Flash-Attention v2 计算词元之间的双向注意力并实现训练与推理的加速;③ 信号解码器,还原调控元件开放性信号。EpiAgent采样全新设计的细胞-调控元件匹配和信号重建任务来进行预训练(图1d),前者判断调控元件是否在细胞语句中开放,后者从细胞表征向量重建完整的调控元件开放性信号。在预训练早期还通过替换语言模型进行模型预热,使模型在不同来源数据上都能稳定学习细胞内调控信息。 EpiAgent作为基础大模型,经过低成本微调即可广泛应用于解决各类单细胞染色质开放性数据下游分析任务。例如,基于细胞表征向量进行非监督细胞聚类,能够使不同细胞类型之间分离更清晰,同一细胞类型内部聚集更紧密。值得关注的是,在与训练数据分布相近的人脑数据上,模型无需微调即可获得优异的聚类效果。在细胞类型的监督注释任务上,模型在经过微调后能够实现细胞类型的高精度分类,特别是在对稀有细胞类型的分类中表现出色。在数据填补任务上,模型在经过微调后能够从细胞表征向量还原所有调控元件的开放状态,有效进行数据去噪和缺失值填补,从而提高下游分析的精度。 图1|EpiAgent原理。a,团队构建了当前最大规模的单细胞染色质开放性数据库Human-scATAC-Corpus。b,细胞语句构建过程。c,EpiAgent模型结构,包括嵌入模块、EpiAgent Transformer及信号解码器。d,全新设计的细胞–调控元件匹配和信号重建预训练任务。 EpiAgent作为细胞语言模型,能够灵活地在细胞语句中引入编码内外源扰动的词元,从而实现细胞扰动响应的准确预测。例如,在细胞词元上附加编码外源刺激的词元,即可准确回归扰动前后差异开放调控元件的强度和方向,预测结果与实验结果高度吻合。在基因扰动响应预测中,通过整合基因本体,模型能够从少量基因扰动实验数据推断其他基因扰动下细胞的染色质开放性信号,预测结果显著优于专门设计的扰动响应预测方法。 近年来细胞图谱建设取得了丰硕的成果,但如何将细胞图谱融入自产实验数据的分析仍然是一个难题。发挥EpiAgent细胞语言模型的灵活性,仅需在细胞词元上附加编码批次效应的词元,即可实现批次效应的矫正,支撑细胞图谱构建。在此基础上,引入互近邻关系将参考细胞图谱标注向实验数据迁移,即可完成实验数据到细胞图谱的映射,从而为整合细胞图谱与实验数据提供了极大便利。 虚拟细胞是当前生物信息学研究的热点,如何通过计算模型对细胞状态进行仿真,实现分子生物学实验的计算模拟,是虚拟细胞应用的关键。清华大学生命基础模型实验室早在2020年就在这一方向进行了前瞻性布局,与美国科学院院士Wing Wong教授共同提出了“数基生命系统”的研究方向。借助细胞语言模型,EpiAgent首次实现了对调控元件进行虚拟敲除的纯计算“数基细胞实验”。如图2所示,仅需在细胞语句中移除调控元件对应的词元,即可模拟对调控元件的敲除,进行敲除后细胞状态的仿真。与 CRISPR实验的对比显示,对基因启动子进行虚拟敲除后,细胞状态与实验结果相似。将该数基细胞实验技术应用于肾透明细胞癌的研究也获得了与文献吻合的结果——对ABCC1、VEGFA及EGLN3启动子的虚拟敲除,能够推动肿瘤细胞向正常细胞演化。这一研究开创了通过数基细胞实验模拟调控元件敲除的研究范式,预期将在药物靶点发现、个性化精准治疗等领域获得广泛应用。 图2|利用EpiAgent进行调控元件虚拟敲除以模拟细胞状态变化。a,EpiAgent通过在细胞语句中移除目标调控元件词元实现虚拟敲除。b-c,与CRISPR 实验的对比显示,对基因启动子进行虚拟敲除后,细胞状态与实验结果相似。d-g,对肾透明细胞癌的研究显示,虚拟敲除ABCC1、VEGFA和EGLN3的启动子能够推动肿瘤细胞向正常细胞演化。 细胞类型的精确标注是单细胞数据分析的基础,目前依赖手动标注的方式存在标准不统一,评判差异大等诸多局限。为克服这些不足,EpiAgent通过在海量标注数据上对基础大模型进行微调,衍生出两个对细胞类型进行自动标注的模型,EpiAgent-B与 EpiAgent-NT,前者适用于对脑相关的细胞进行自动标注,后者则适用于其他组织器官。这两个模型可以直接使用,不需要再进行微调,从而开创了细胞类型自动标注的“零样例”学习新范式。 EpiAgent通过细胞压缩语言模型,将细胞内由百万染色质开放性区域刻画的表观基因组建模为细胞语句,在统一语境中用统一方法解析复杂的基因调控规律,从而适配纷繁复杂的下游分析任务,其“统一建模,广泛应用”的全新研究范式,打破了以往生物信息学研究“一个问题,一个模型”的桎梏,为单细胞表观基因组学提供了全新的统一解决方案,为开展扰动响应预测、调控元件虚拟敲除等研究提供了行之有效的手段。这一开创性成果不仅是国际上第一个单细胞表观组基础大模型,更建立了面向药物靶点发现、个性化精准治疗等应用的数基细胞实验新范式,从而开辟了基于表观基因组的虚拟细胞研究。 EpiAgent于2025年9月25日发表在顶级期刊Nature Methods。论文第一作者是清华大学自动化系2020级博士生陈晓阳,通信作者是清华大学自动化系长聘教授江瑞,其他作者包括:清华大学自动化系李可伊、崔雪建、王子安、江群、林嘉成、李震、高子靖和闾海荣。论文得到了国家重点研发计划(2023YFF1204802、2025YFC3409300、2022YFF1202400和2021YFF1200902)、国家自然科学基金(62273194),以及北京市自然科学基金(L242026)的资助。 论文信息:Chen, X., Li, K., Cui, X. et al. EpiAgent: foundation model for single-cell epigenomics. Nature Methods (2025). https://doi.org/10.1038/s41592-025-02822-z