Nat Methods:超越传统ST平台!iSCALE实现大组织切片的高分辨率空间基因表达预测和组织注释
时间:2025-09-25 15:50:09 热度:37.1℃ 作者:网络
革命性技术空间转录组学(ST)能在保留空间背景信息的前提下分析基因表达,彻底改变了我们对复杂组织内细胞组织、功能和相互作用的理解。随着ST技术的迭代创新,其在肿瘤学、免疫学、发育生物学等领域展现出巨大的潜力,催生出一系列高质量的科研成果。但现有的ST平台仍存在成本高昂、耗时过长、分辨率不足、基因覆盖范围有限以及固有捕获区域较小等不足,限制了其在临床领域的广泛应用,尤其是在人类研究中常见的大组织样本中。
近日,美国宾夕法尼亚大学Mingyao Li团队及合作者提出了新的机器学习框架iSCALE,这是一种预测大规模、细胞分辨率基因表达图谱的方法,可在超过当前ST平台捕获区域的样本中自动注释细胞水平的组织结构。该成果已发表在Nature Methods上,文章题为“Scaling up spatial transcriptomics for large-sized tissues: uncovering cellular-level tissue architecture beyond conventional platforms with iSCALE”。性能评估显示,iSCALE能够在整个大组织切片上进行全面的基因表达预测和组织注释,包括没有直接基因表达检测的区域,揭示了常规ST实验无法检测到的病变相关细胞特征。同时,该研究结果证明了iSCALE在分析大规模组织样本方面的实用性,能够实现无偏注释、解析细胞类型组成、绘制细胞微环境和揭示超出标准ST分析或常规组织病理学评估范围的空间特征,为疾病研究提供新见解。

主要研究内容
临床常规使用的苏木精和伊红(H&E)染色是病理组织诊断的“金标准”,其标准切片尺寸可达25mm×75mm,远超现有ST平台的捕获面积。为此,研究团队开发了iSCALE计算框架,以预测大组织切片H&E图像的基因表达。与依赖于单个ST捕获进行预测的方法不同,iSCALE通过将单个切片映射到完整的组织图像并整合多个ST捕获的信息,从而实现空间连续的基因表达预测。
iSCALE首先从组织样本中选择几个区域生成小的训练ST捕获集(子捕获),对其进行基因表达数据空间聚类,与H&E图像对齐,并将基因表达和空间信息整合在对齐的子捕获中。随后,iSCALE从H&E图像中提取整体和局部组织结构信息,利用神经网络学习子捕获中的组织图像特征与基因表达之间的关系。由此产生的模型可预测整个H&E图像中每个8μm×8μm超像素(约为单个细胞大小)的基因表达,获得整个组织切片的高分辨率基因表达谱。最后,iSCALE进一步用细胞类型注释H&E图像中的每个超像素,并识别每个组织区域中富集的细胞类型,以细胞分辨率详细表征组织结构。总之,iSCALE通过对不同ST切片的信息进行对齐和组合,利用大尺寸的H&E图像进行大规模组织区域的基因表达预测及组织注释,包括没有直接基因表达检测的区域。
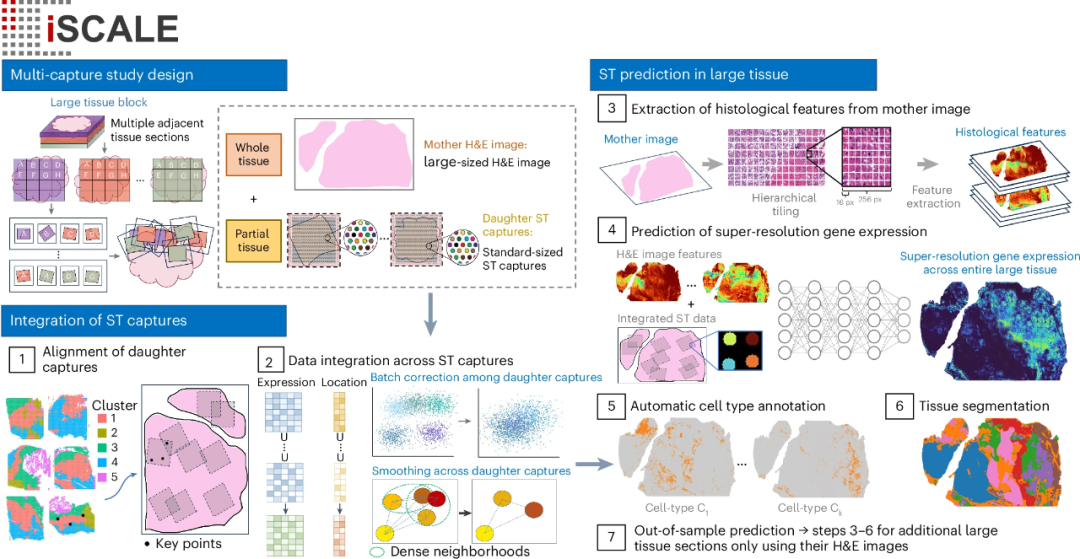
图1. iSCALE工作流概述。
研究团队通过基准实验评估了iSCALE的有效性。使用来自10x Xenium平台的大型胃癌组织切片(12 mm×24 mm,377个基因)的真实单细胞基因表达数据集,模拟了真实大组织研究中的条件,并使用iStar16和RedeHist19预测基因表达来进行对比。(图2)结果显示,iSCALE的组织分割与病理学家的手工注释高度相似,并成功识别了关键的组织结构,包括肿瘤、肿瘤浸润间质、粘膜和三级淋巴结构等。相比之下,iStar和RedeHist的组织分割存在明显差异。值得关注的是,iSCALE检测到与胃癌预后相关的印戒细胞区域,这是其他两个方法无法实现的。
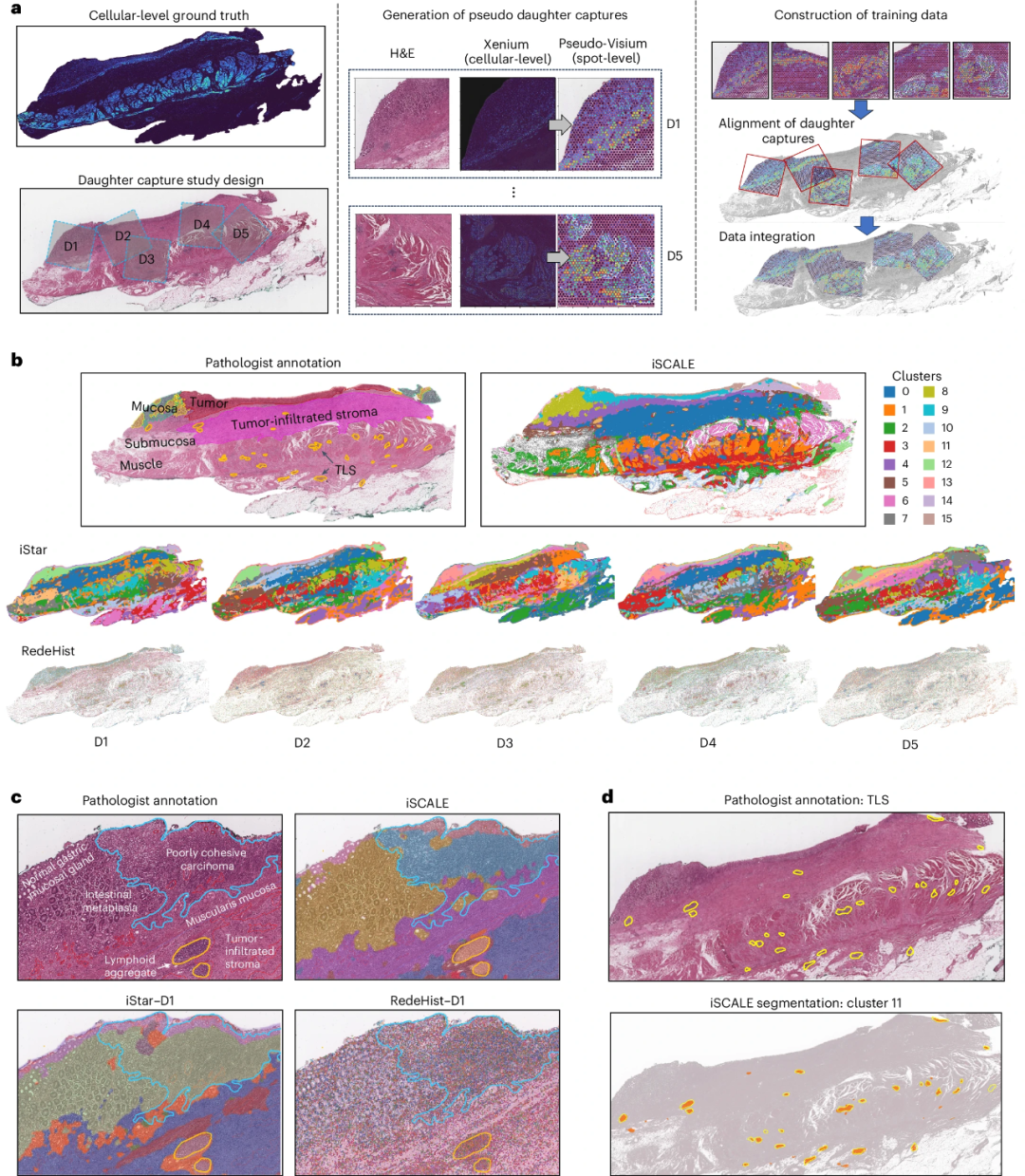
图2.利用胃癌组织切片评估iSCALE。
研究团队比较了两个版本的iSCALE:iSCALE- seq(使用伪Visium数据进行训练)和iSCALE- img(使用Xenium数据进行训练),分析了前100个高可变基因(图3)。评估显示,iSCALE-Seq在所有评估指标上都优于iStar,性能与iSCALE-Img相似。iSCALE-Seq在32µm×32µm的空间分辨率下,大约50%的基因获得了大于0.45的相关系数。在可变基因鉴定中,iSCALE的表现更强。所有基因数据结果表明,与iStar相比,iSCALE具有稳健性和一致性的优越性能。将iSCALE和iStar预测的基因表达与Xenium检测的真实单细胞表达进行可视化比较,发现在所有被检测的基因中,iSCALE的预测与真实基因表达最相似。
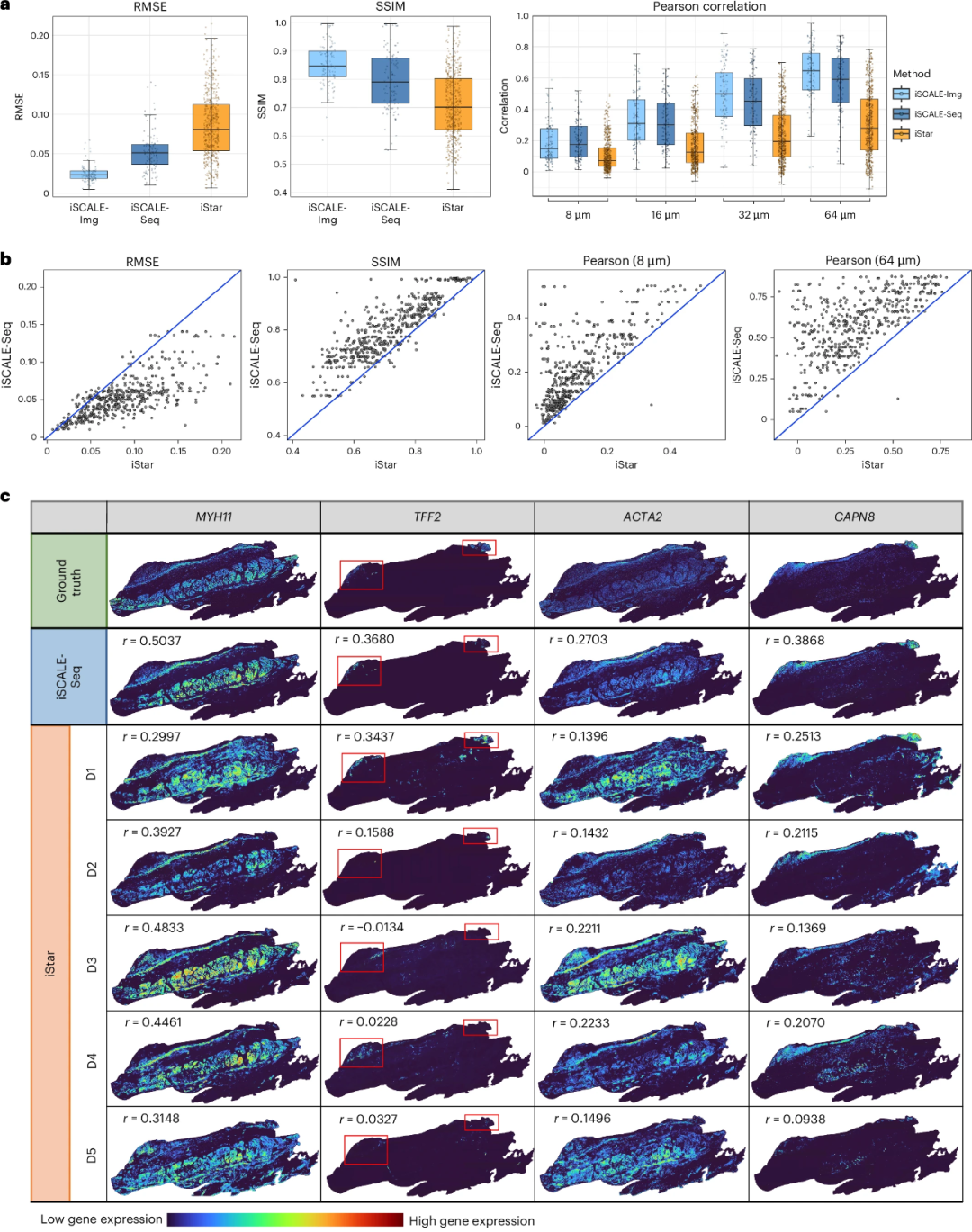
图3.胃癌组织切片中iSCALE预测准确性。
此外,研究还评估了iSCALE在样本外预测方面的表现,样本为来自不同受试者的正常胃组织切片并使用10x Xenium平台分析,且仅包含没有可用基因表达数据的H&E图像。结果显示,iSCALE样本外预测结果与病理学家注释的分割结果高度接近。
研究团队进一步将iSCALE方法应用于来自多发性硬化症(MS)患者的死后人脑生成的数据。iSCALE预测了整个H&E图像的超分辨率基因表达,包括2,013,871个超像素。这些超分辨率基因表达数据使iSCALE能够分割组织并识别与病理学家手工注释密切匹配的区域(图4)。iSCALE成功地在整个图像上连续检测到白质慢性病变核心、小胶质细胞激活增加的边缘、外观正常的白质和外观正常的灰质。
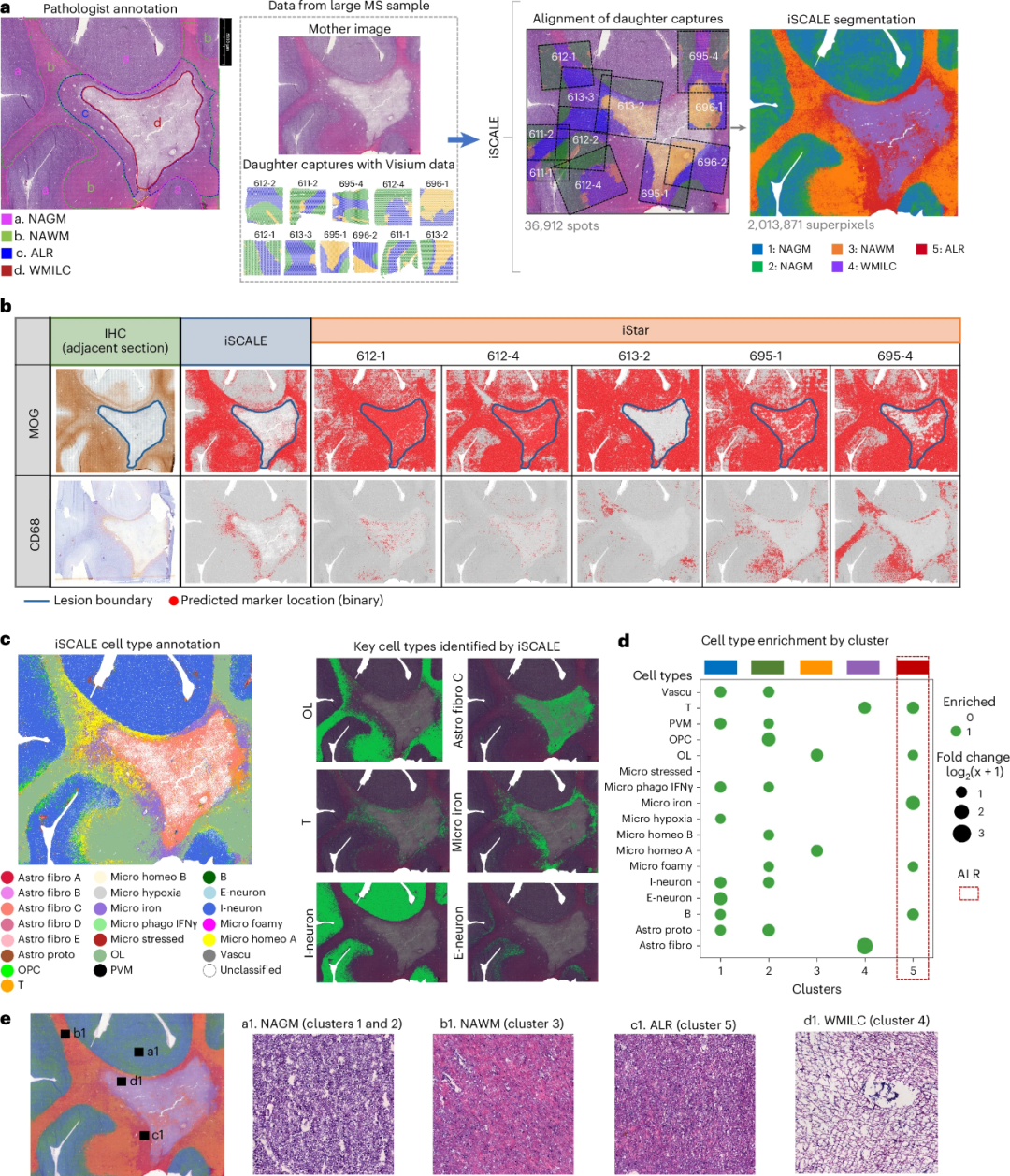
图4:将iSCALE应用于人类MS脑样本。
结 语
该研究开发的iSCALE可用于快速基因表达预测、细胞类型注释和组织分割,有效地处理大型组织样本,超过了传统ST平台的能力。与目前最先进的方法相比,iSCALE能够为空间基因表达分析提供更全面和可扩展的方法。H&E图像可广泛获取,且成本效益高。相信随着更多的ST数据可用,iSCALE应用将逐步推广。此外,iSCALE的样本外预测能力可以大大降低实验成本,使大规模ST研究更加可行。总之,iSCALE有助于提高对细胞水平的空间组织结构的理解,为多种人类疾病提供有价值的见解。
原文信息:
Schroeder, A., Loth, M.L., Luo, C. et al. Scaling up spatial transcriptomics for large-sized tissues: uncovering cellular-level tissue architecture beyond conventional platforms with iSCALE. Nat Methods 22, 1911–1922 (2025). https://doi.org/10.1038/s41592-025-02770-8


