Nat Methods:突破分辨率桎梏!GHIST框架首次从常规病理图像中直接预测单细胞空间基因表达
时间:2025-09-25 15:50:18 热度:37.1℃ 作者:网络
空间分辨转录组学(SRT)技术能够提供基因表达的空间定位信息,不仅革新人们对多细胞生物系统的理解,还能助力改善癌症等复杂疾病关键临床结局的预测,但这类技术成本高昂,限制了其广泛应用。
相比之下,H&E病理组织图像成本低且易获取,其蕴含丰富的细胞形态与组织结构信息,成为预测基因表达的潜在替代来源。但现有深度学习方法仅能实现spot级基因表达预测,还存在分辨率不足等多种问题。从spot级到单细胞级预测面临更大挑战:需处理海量表达谱、相似形态细胞难区分、基因表达受邻近细胞影响,且细胞形态、基因表达等多层面关系尚未被充分利用。
为解决上述难题,悉尼大学精密数据科学中心的研究团队提出了一种名为GHIST的深度学习框架,能够从H&E组织图像中预测单细胞水平空间基因表达(SGE)。该框架利用多预测组件,整合细胞类型、细胞核形态、邻域组成和单细胞表达等多层次生物信息;并借助多任务学习架构和多损失函数,协同捕捉不同类型信息的相互依赖关系,从而提升SGE预测效果。经验证,GHIST展示了在不同空间分辨率的灵活性和优越性能。总之,GHIST提供了一种创新的计算策略,丰富了现有病理数据库的空间组学维度,为生物标志物发现及疾病机制研究提供了低成本、高效率的解决方案。

GHIST是一种多任务深度学习模型,通过将H&E图像映射到图像中单个细胞内数百个基因的表达,并提供细胞空间定位,从而实现单细胞级SGE预测;本质上,该方法是将H&E图像映射为SGE图像集合(通道数等于基因数,像素强度对应细胞内单个基因的总表达量)。训练时,GHIST使用配对H&E图像及SST数据,并利用单细胞RNA-seq数据优化预测;训练完成后,该模型仅需输入H&E图像即可输出空间单细胞基因表达矩阵,无需SST数据和额外空间组学信息支持。
GHIST的核心创新在于其多任务架构整合了四个层次的生物学信息:细胞类型、细胞核形态、邻域细胞组成和基因表达。模型通过四个对应的预测头协同学习这些信息之间的依赖关系,并借助损失函数增强跨任务信息交互。
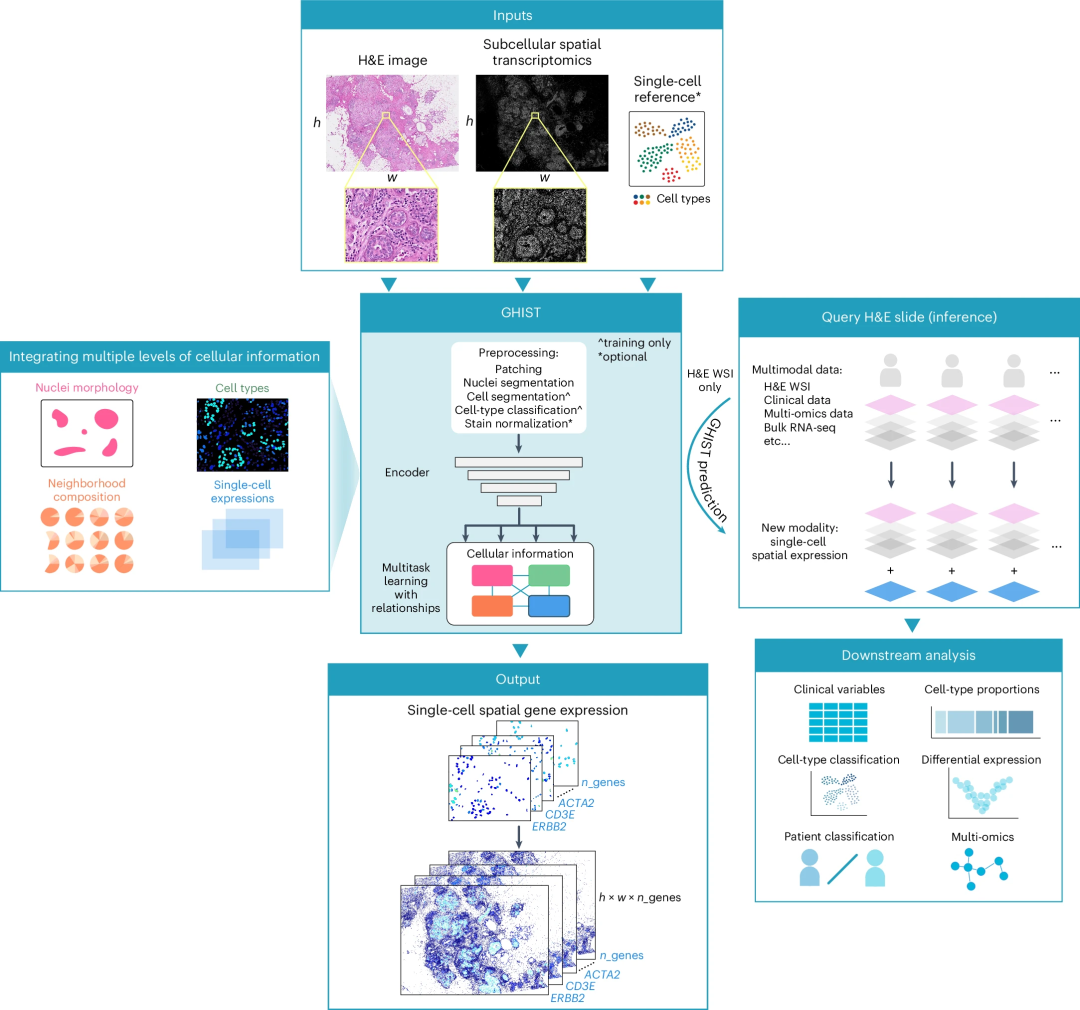
图1. GHIST框架
研究团队采用细胞类型比例、空间可变基因(SVGs)、相关性等指标,通过比较预测表达与真实基因表达,评估了GHIST预测的准确性。在两张乳腺癌H&E图像中,GHIST预测的细胞类型空间分布与SST真实数据高度吻合;多分类预测(8类细胞)中,准确率分别达0.75和0.66。
该模型对SVG的预测表现尤为突出,预测值与真实值高度一致,前20和前50 SVG的中位皮尔逊相关系数(PCC)分别达0.7和0.6;乳腺癌相关基因SCD、FOXA1、EPCAM等预测值与真实值也高度相关。此外,在肺癌腺癌和黑色素瘤数据集中,GHIST预测的细胞类型比例与真实值之间相关性分别达0.97和0.92,证明其跨癌症类型的适用性。
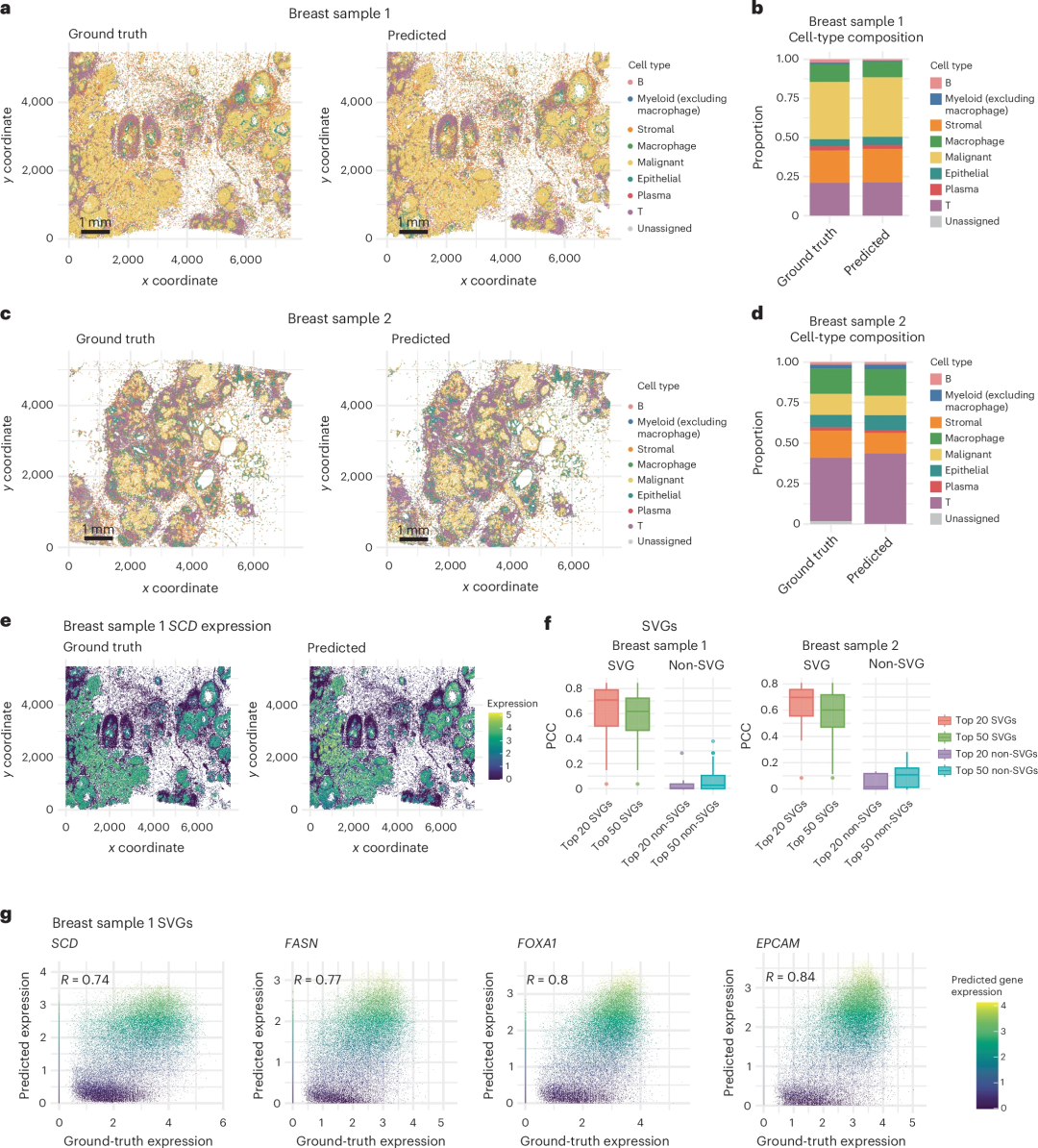
图2. GHIST对两张乳腺癌H&E图像的预测
除单细胞级SGE外,GHIST还可扩展应用于预测spot级SGE。在人类HER2+乳腺癌数据集(HER2ST)中,GHIST的表现优于ST-Net、Hist2ST、DeepPT等现有方法,其在所有基因上的平均PCC达0.16、结构相似性指数(SSIM)达0.10。
对于生物学意义显著的基因(如GNAS、FASN)、高变基因(HVGs)和SVGs,GHIST也表现出最高相关性;前10% HVG和前20名SVG的PCC分别达0.20 和0.27,SSIM分别达0.17和0.26,全面优于同类工具。与iStar方法相比,降低分辨率后GHIST仍在多项指标中显著领先,且能避免无信号基因的虚假相关性。
进一步,基于TCGA-BRCA数据集的生存分析显示,利用GHIST预测表达值构建的多变量Cox回归模型在交叉验证中取得最高C指数(0.57),能够显著区分高风险与低风险患者,这表明GHIST预测结果具有转化应用潜力,可用于临床结局预测。
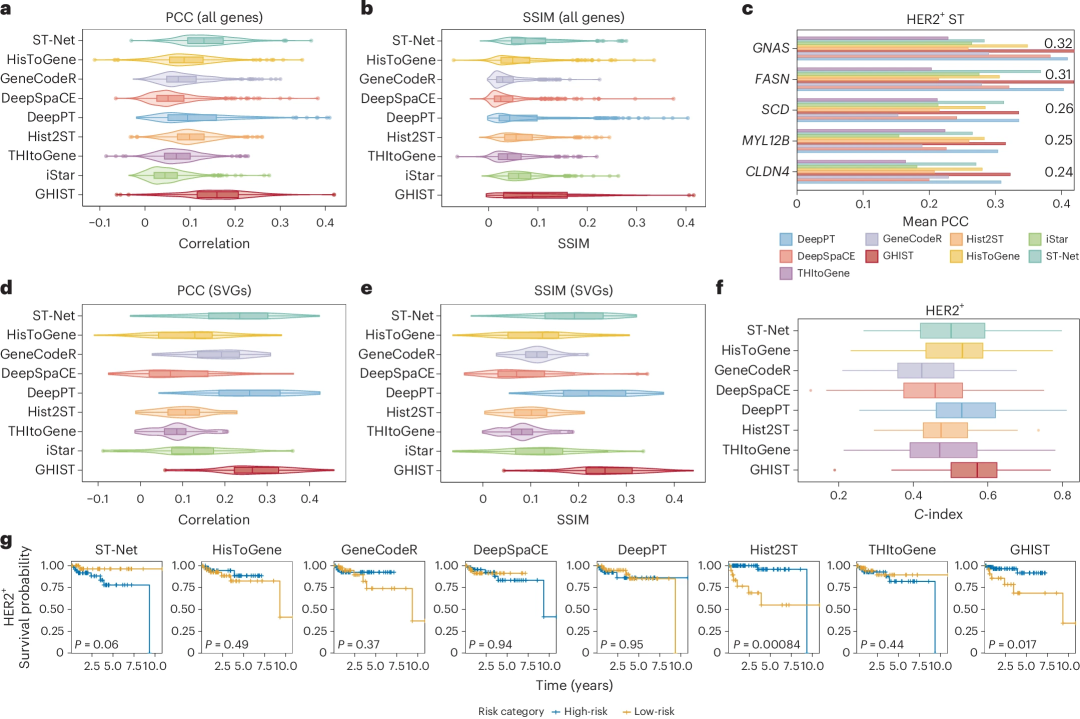
图3. GHIST与其他方法的性能比较
为评估GHIST的实际应用效果,研究团队将其直接应用于两个外部数据集:一个是TCGA-BRCA数据集,包含92例HER2+、461例管腔型样本,以及一个包含44例患者的内部乳腺癌组织数据集。以H&E图像作为输入,GHIST成功预测了单个细胞中280个基因的表达。
结果显示,恶性细胞标志物EPCAM与肿瘤微环境高表达基因SFRP4的空间分布符合预期,恶性细胞和基质细胞的总体比例与生物学认知一致。2名恶性细胞预测极少的患者,经病理医生评估确认存在H&E染色质量缺陷。在内部数据中,GHIST预测结果进一步揭示浸润区域较原位区域具有更高的免疫浸润程度,与文献报道一致,验证了预测结果的生物学可靠性。
GHIST为TCGA样本生成了空间单细胞表达图谱,从而为TCGA多组学数据集增添了新的分析维度,可通过多项下游分析展现潜力。在生存分析中,利用GHIST提取的细胞类型信息与SGE模式能够有效区分患者风险群体。在HER2队列中,细胞类型特异基因比例与TCGA批量RNA-seq数据的区分风险能力相当;在样本量更大的管腔亚型队列中,空间指标(如邻近相关性和Moran's I)表现出更高的预后显著性。
在TCGA HER2队列中,基于细胞类型特异性信息可揭示ER/PR阳性人群的患者异质性:其中高表达LPL、TIMP4等基因的群体预后更好。此外,基于空间模式特征(邻近相关性等)可有效区分不同亚群(准确度0.66),并识别出与生存相关的空间模式基因如TNFRSF17、CD19等。
此外,研究团队还揭示了体细胞拷贝数变异(SCNA)与基因空间模式的关联,在8号及17号染色体上发现了与癌症风险相关的空间调控热点区域,其中TOP2A拷贝数变异影响显著。上述发现凸显了GHIST在拓展空间多组学分析方面的能力。
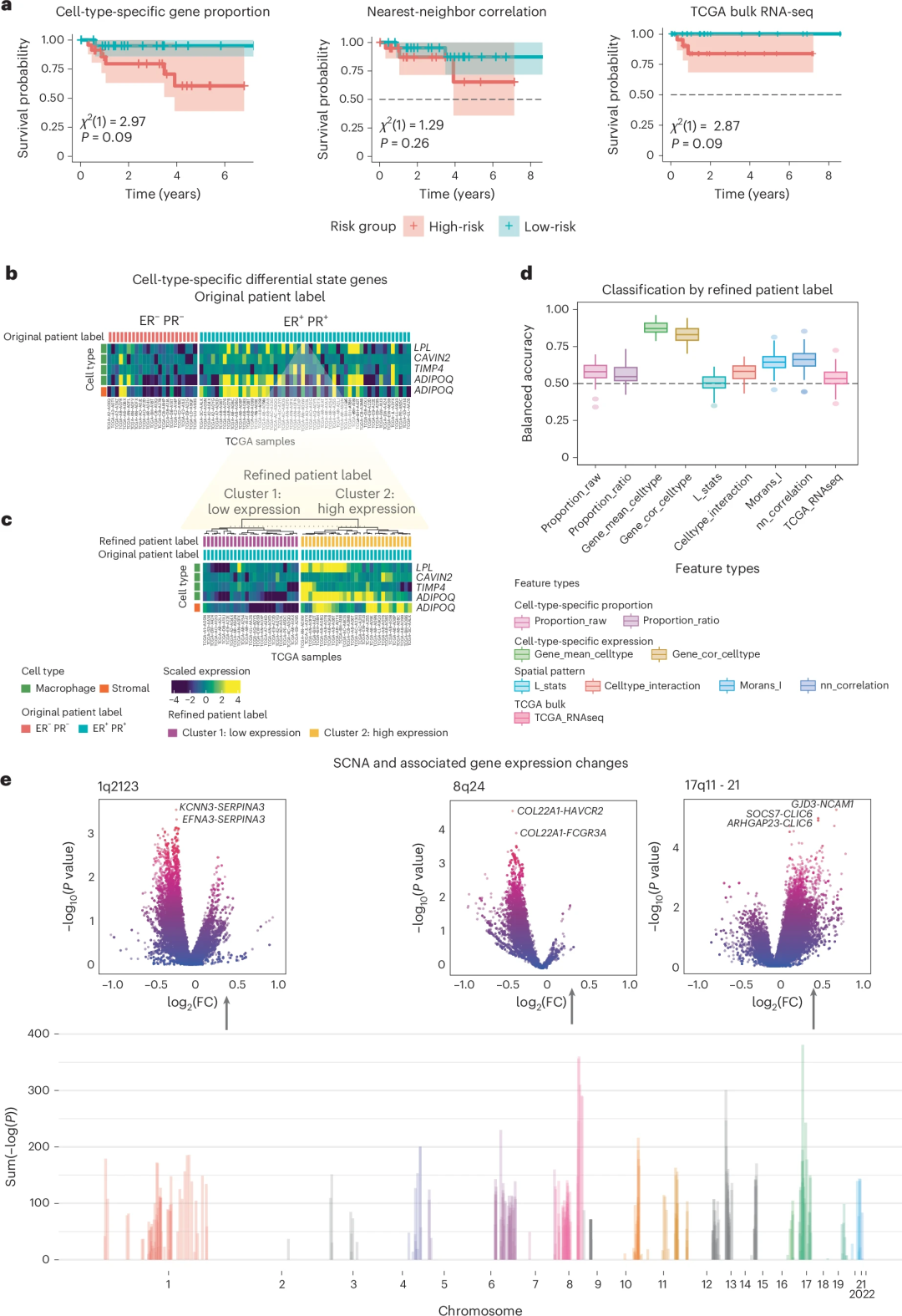
图5. GHIST在创建多视角分析方面的潜力
综上所述,GHIST首次实现从常规H&E图像直接预测单细胞水平空间基因表达,突破了现有方法的分辨率与成本瓶颈。该框架提供了一种低成本、高通量的计算策略,为海量临床病理数据赋予空间组学维度,显著丰富TCGA等数据库的多组学分析能力;支持多种下游分析,推动了精准医学和空间多组学的发展。
参考文献:
Fu, X., Cao, Y., Bian, B. et al. Spatial gene expression at single-cell resolution from histology using deep learning with GHIST. Nat Methods (2025). https://doi.org/10.1038/s41592-025-02795-z


