从 Exomiser 到 Genomiser:基于 985 例罕见病患者数据,探索全基因组编码与非编码变异诊断性能优化
时间:2025-09-28 12:13:21 热度:37.1℃ 作者:网络
随着全基因组测序技术在罕见病诊断中的广泛应用,表型驱动的变异优先排序工具成为提升诊断效率的关键环节,Genomiser作为Exomiser的扩展版本,能够同时分析编码区和非编码区变异,但其在真实世界全基因组数据中的性能尚未得到系统评估。香港基因组项目作为香港首个大规模基因组测序计划,旨在推动基因组医学的发展,项目初期已将Exomiser纳入诊断流程,但随着测序技术的进步和数据分析需求的提升,亟需对更全面的变异类型进行有效解读。非编码变异在罕见病中占据一定比例,但由于其功能注释的复杂性和现有预测工具的局限性,临床应用中常面临假阳性高、优先级混乱等问题。本研究基于香港基因组项目的实际数据,旨在通过参数优化提升Genomiser在临床诊断中的敏感性和可靠性,探索其在全基因组层面识别致病性非编码变异的潜力。

研究纳入了香港基因组项目中985名已披露全基因组测序结果的罕见病患者,其中207例为经美国医学遗传学与基因组学学院/分子病理学协会指南确认的阳性病例,包括14例由非编码变异致病的个案,其余778例为初始报告阴性的病例。所有患者的表型信息均采用人类表型本体术语标准化录入,并通过内部临床门户系统统一管理。测序数据采用Illumina短读长测序平台生成,平均覆盖深度≥30×,并通过基于GRCh38参考基因组的二级分析流程进行比对和变异检测。在Genomiser参数优化过程中,研究者扩展了基因区间过滤器至转录本边界上下游2000碱基对,以涵盖启动子、增强子及深度内含子区域,并将次要等位基因频率阈值调整为3%,以兼容外显率降低的致病变异。此外,工具运行中启用了SpliceAI和ReMM等非编码变异预测模块,并针对编码变异保留了REVEL、MVP和AlphaMissense等多种致病性预测源。
在敏感性评估中,研究者首先比较了默认参数下Exomiser与Genomiser的性能表现。结果显示,在Top-3候选变异中,Exomiser的敏感性为72.14%,而Genomiser仅为70.15%,在Top-5排名中二者分别为80.60%和74.63%,表明Genomiser在默认设置下整体敏感性较低。进一步分析发现,Genomiser中ReMM评分较高的非编码变异频繁出现在优先级前列,导致真实致病性编码或非编码变异排名被压低,例如在一例智力障碍患者中,PHIP基因的致病变异因被六个ReMM评分高于0.9的非编码变异超越而仅排在第七位。这一现象在表型非特异性或遗传异质性高的病例中尤为突出,凸显了ReMM评分在未经过滤时引入的噪声问题。
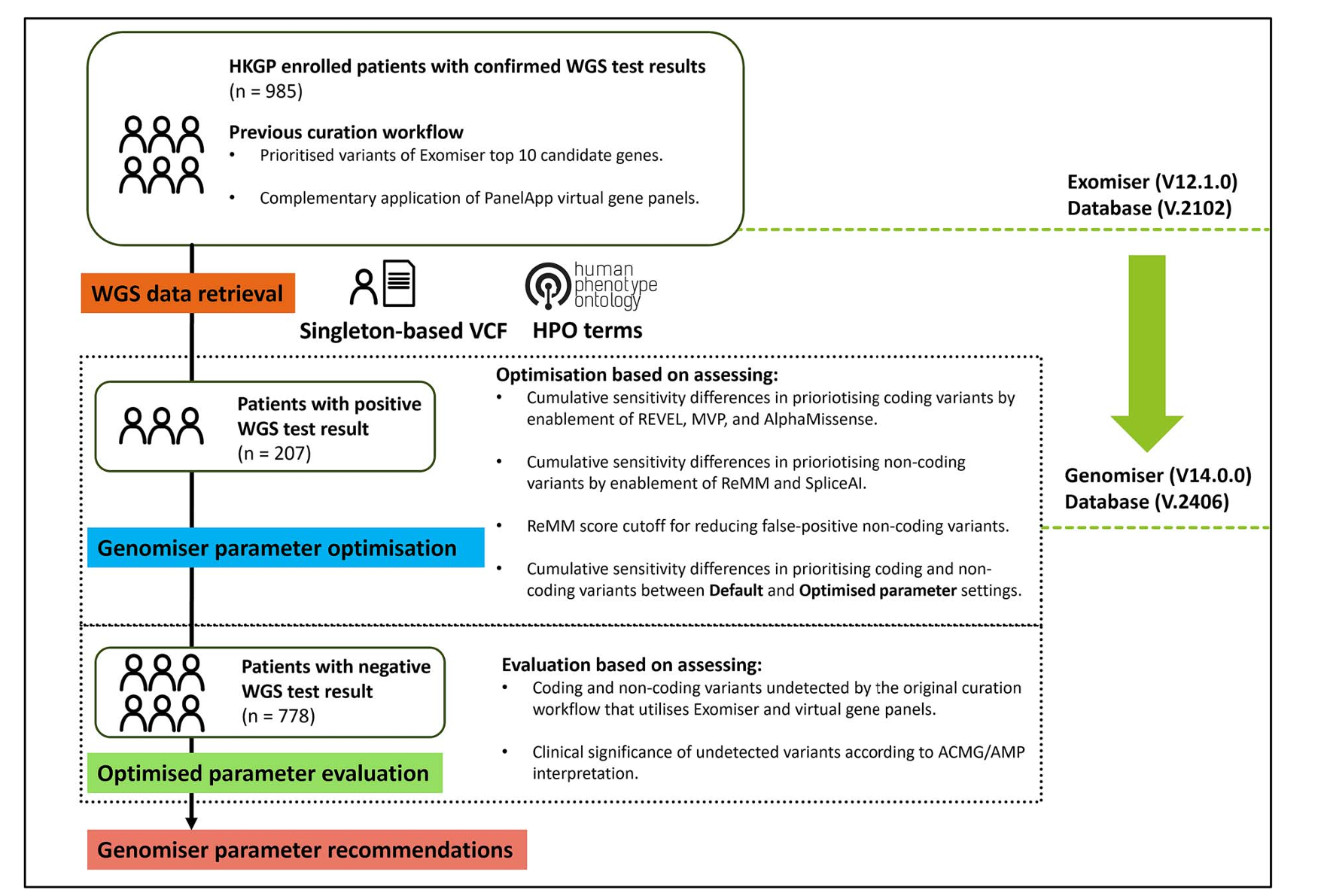
图:研究流程概览
为优化Genomiser的性能,研究者重点调整了ReMM评分的过滤阈值。通过比较0.963、0.97、0.98和0.99四个阈值发现,随着阈值提高,Genomiser的敏感性逐步上升,当ReMM阈值设为0.963时,Genomiser在Top-15候选变异中的敏感性达到92.54%,优于Exomiser的89.55%。而当阈值升至0.99时,敏感性虽进一步提高至96.02%,但可能因过滤过于严格而漏掉部分真实致病性非编码变异。基于敏感性-特异性平衡的考量,最终选定0.963作为ReMM的优化阈值。与此同时,编码变异预测模块中同时启用REVEL、MVP和AlphaMissense时,敏感性较单一REVEL有所提升,因此优化参数中保留了这三项工具。
将优化后的参数应用于778例初始阴性病例的重新分析中,Genomiser共识别出20个与患者表型高度匹配的深度内含子变异,这些变异均经SpliceAI预测具有剪接影响,并通过MaxEntScan进一步验证。其中5个变异依据ACMG/AMP指南可直接判定为致病性或可能致病性,从而为这些患者提供了明确的分子诊断。例如,一例疑似Dravet综合征的患者在经历SCN1A基因多轮检测均为阴性后,通过优化后的Genomiser在其第八优先级发现一个de novo深度内含子变异(c.2415+431C>G),该变异经剪接预测工具确认可产生隐秘剪接位点,最终被归类为可能致病性,为患者后续治疗提供了关键依据。其余15个变异因缺乏足够表型特异性或功能验证数据,暂被归类为临床意义未明变异,但为进一步的功能研究和表型再评估指明了方向。
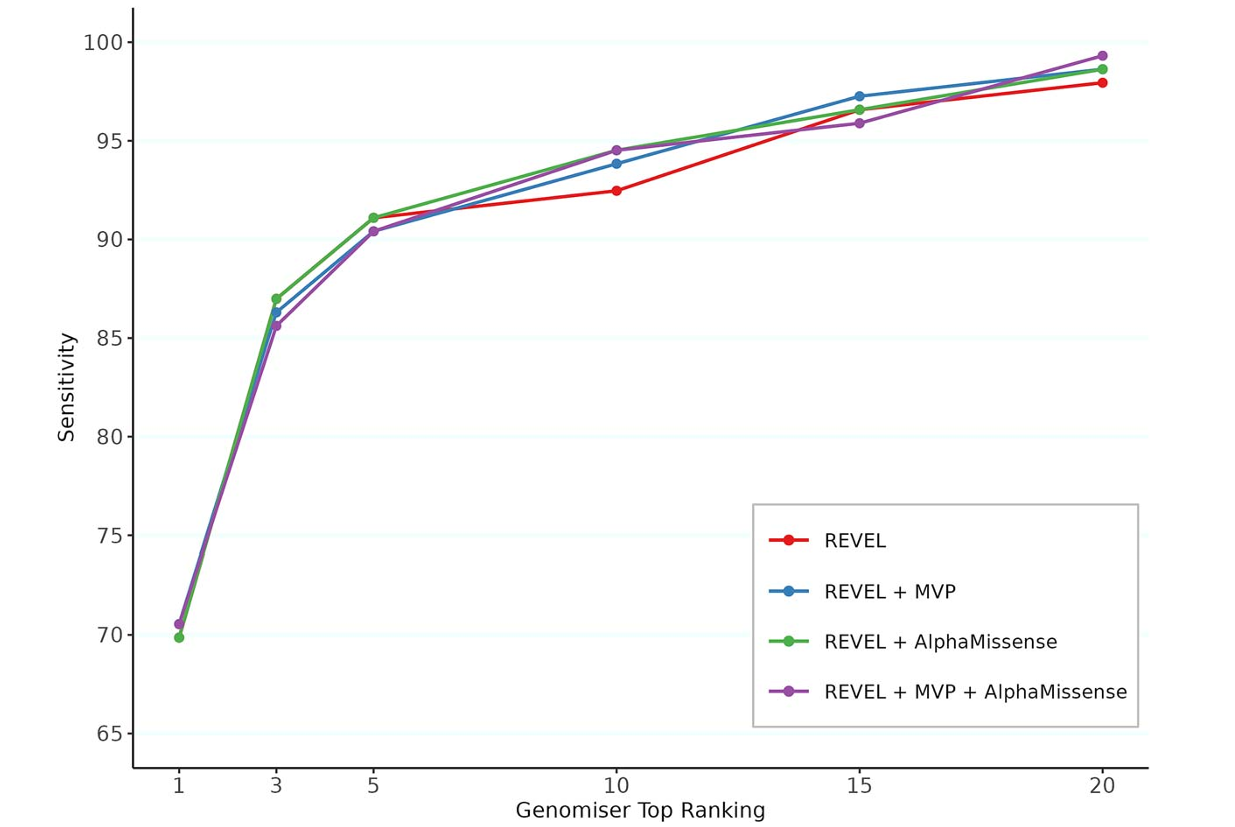
图:Genomiser使用不同编码变异致病性来源组合的累积敏感性分布曲线
本研究通过系统化的参数优化,显著提升了Genomiser在真实世界全基因组数据中的诊断效能,使其在兼顾编码变异分析的同时,有效拓展了对非编码变异的识别能力。优化后的流程仅需审阅Top-15候选变异即可达到92.54%的敏感性,极大提高了临床诊断的工作效率。此外,研究中采用的SpliceAI、REVEL等工具均为ClinGen专家组推荐,保证了变异注释的规范性和可重复性。尽管非编码变异的数据稀缺性和预测工具的内在偏差仍是当前挑战,但通过表型驱动的优先级排序和合理的参数设置,Genomiser有望成为罕见病全基因组分析中的核心工具。香港基因组项目未来还将结合多组学数据对本次发现的临床意义未明变异进行功能验证,进一步推动非编码变异在精准医疗中的应用。
原始出处:
Xi, A. M. C., Yeung, D. L. H., Ma, W., Ying, D., Tong, A. H. Y., Or, D., Hue, S. P. Y., Hong Kong Genome Project, Chu, A. T.-W., & Chung, B. H.-Y. (2025). Official and data-driven optimization of Genomiser for rare disease patients: experience from the Hong Kong Genome Project. *Briefings in Bioinformatics*, 26(5), bbaf475. https://doi.org/10.1093/bib/bbaf475
本文相关学术信息由梅斯医学提供,基于自主研发的人工智能学术机器人完成翻译后邀请临床医师进行再次校对。如有内容上的不准确请留言给我们。


